introduktion:
mikrober är för små för att ses av blotta ögat; de kan överleva under förhållanden som många skulle tro är olösliga som den anaeroba miljön i våmmen av kor, varma källor och kalla Antarktiska vatten (Vad är mikrober, 2010). Miljontals mikrober lever både på och i människokroppen och kan båda hjälpa oss att överleva eller göra oss sjuka, mindre än 1% av bakterierna orsakar sjukdom (Vad är mikrober, 2010).,
mikrobiomen i näshålan består huvudsakligen av fylaaktinobakterier, Firmicutes och Proteobakterier (Bassis et al. 2014). Mikrobiomen i näshålan kan också förändras som svar på miljöfaktorer som geografisk plats och hygien (Rawis et al. 2019).
Micrococcus luteus finns på många ställen, inklusive hud, jord, damm, vatten, luft, mun, mucosae, orofarynx och övre luftvägarna hos människor (Wikipedia, Micrococcus luteus, 2019). Det är en gram-positiv, coccusformad mikrobe och innehåller katalas. Denna mikrobe bildar stora, runda kolonier., Det kan lätt misstas för stafylokocker, eftersom de är mycket lika morfologiskt och fysiologiskt (Wikipedia, Staphlyokocker Aureus).
mitt mål i detta experiment var att isolera, karakterisera och identifiera en bakteriell koloni som uppstod från ett prov som togs från min rumskamrats näsa. Jag hypoteserade att det skulle vara en bakterie som vanligen finns i näshålorna och sannolikt från ovan nämnda phyla, så det skulle sannolikt göra bäst att leva i en aerob, fuktig och varm miljö.
metoder:
Jag valde att prova bakterier inifrån min rumskamrats näsa., För att prova använde jag sterila bomullspinne och strödde dem på TSA-plattor. Jag höll plattan vid rumstemperatur i 7 dagar och valde sedan en koloni för att rena med hjälp av pure culture streak plate-metoden. Jag upprepade denna process tre gånger för att ytterligare rena kolonin. När kulturen ansågs ren nog inokulerade jag ett slantrör.
jag utförde många tester för att ta reda på kolonins morfologi och fysiologi., För att bestämma fysiologiska egenskaper hos kulturen som cellform, arrangemang och om det var gram-positivt eller negativt, vilket hjälper till att bestämma cellväggstypen för mikroben, utförde jag en gram-fläck. Jag använde en oxidastestremsa och vatten för att bestämma om cytokrom C-oxidas var närvarande och utförde ett katalastest för att bestämma om katalas var närvarande. Jag gjorde också ett vätsketioglykolattest för att bestämma bakteriens syreklass., Jag växte mina bakterier på en eosin metylenblått (EMB) platta för att se om det kunde jäsa laktos och om det kunde växa med metylenblått som väljer för gramnegativa bakterier. Jag växte också den i en MacConkey (MAC) platta för att se om det kunde jäsa laktos och om det kunde växa med både kristallviolett och gallsalter för att ytterligare bekräfta om det var gram negativt eller positivt. Jag använde ett API Strep-test för att bestämma mer av sockerarterna som bakterierna kunde jäsa.
Jag växte min isolerade i Tryptisk Sojabuljong (TSB) i en vecka för att förbereda sig för DNA-extraktion., Jag extraherade DNA med hjälp av PowerSoil DNA kit (Tillverkad av Qiagen) enligt tillverkarens instruktioner. Provet sekvenserades sedan med Illumina MiSeq-tekniken i UAF: s DNA-Kärnlabb. Jag använde PATRIC programvara för att utföra en metagenome binning och att tilldela en taxonomi till bakterierna.
resultat:
kolonin tog 16 dagar att renas. Gramfläcken av denna mikrobe visade att den är gram positiv eftersom den färgade lila. Denna mikrobe är coccusformad och bildar sig i tetrader. Kolonin bildas som en gul, glänsande rund klump., Katalas-och oxidastesterna kom upp negativa, eftersom katalastestet inte bildade bubblor, och oxidastestet såg ingen färgförändring. Oxidastestet för att se om mikroben innehåller cytokrom C-oxidas. Katalasprovet testar för att se om mikroben innehåller katalas. Vätsketestet tioglycallate visade att bakterierna var en obligat aerobe eftersom tillväxten koncentrerades på toppen av röret i den rosa regionen. MacConkey-agar visade mycket liten tillväxt och hade ingen färgförändring, vilket indikerar att mikroben var gram-positiv och inte en fermentor., EMB-agar visade ingen tillväxt eller färgförändring, vilket också indikerar att mikroben var gram-positiv och en icke-fermentor.
den taxonomiska tilldelningen av denna mikrobe var micrococcus luteus eftersom det var den enda bin som PATRIC gav. Den hade 27 372 contigs i församlingen. Det har flera antibiotikaresistensgener inklusive dihydropteroatsyntas, glycerofosforyldiesterfosfodiesteras och SSU ribosomala proteiner.
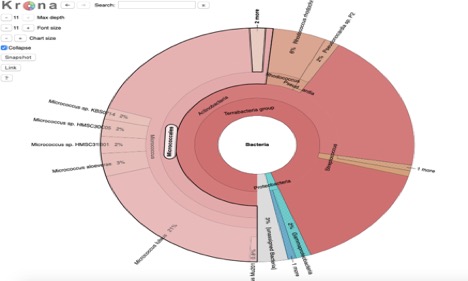 Figur 1. Krondiagram av mikrob visar bakterieklasser som tros vara närvarande i provet.
Figur 1. Krondiagram av mikrob visar bakterieklasser som tros vara närvarande i provet.
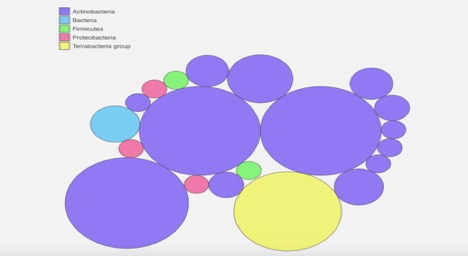 Figur 2., Kaiju webbserver metagenome binning analys diagram. Det visar att provet innehåller bakterier från Terrabacteria grupp. Det är mestadels aktinobakterier, men vissa Proteobakterier och Firmicules finns också i provet.
Figur 2., Kaiju webbserver metagenome binning analys diagram. Det visar att provet innehåller bakterier från Terrabacteria grupp. Det är mestadels aktinobakterier, men vissa Proteobakterier och Firmicules finns också i provet.
kaiju metagenome binning visar att mikrobeprovet inte är helt rent (Figur 2). Det visar att det är mestadels aktinobakterier ,med vissa firmicules och Proteobakterier blandade i (Figur 2). Detta stämmer överens med PATRIC metagenome binning som också visade vissa föroreningar (Figur 1).,
diskussion:
eftersom mikroben är gram-positiv betyder det att den har ett stort peptidoglykanlager och saknar ett lipopolysackaridlager. MacConkey agar är selektiv för gramnegativ, varför min mikrobe inte visade mycket tillväxt på den, och eftersom den inte ändrade färger betyder det att den inte jäste laktosen. EMB-plattan är också selektiv för gramnegativa bakterier vilket förmodligen är anledningen till att bakterierna inte växte på den. Syreklassen i mikroben, obligate aerobe, matchar de förutsägelser jag hade gjort om det eftersom bakterierna ursprungligen hämtades i en näsborre., Wikipedia säger också att Micrococcus luteus är en obligatorisk aerobe, som säkerhetskopierar vad mina resultat visar (2019).
oxidastestresultaten tyder på att mikroben inte innehåller oxidas, trots vad metagenomebinningstestet visade. Katalastestet visade också att mikroben inte har katalas, trots det metagenomiska binningstestet som tyder på det. Dessa skillnader kan bero på mänskliga fel, unpure kultur eller en gammal agar platta., API-testremsorna brist på resultat tyder på att jag använde fel testremsa, jag behövde förmodligen använda Staph-testet istället för Strep-testet, eftersom Strep-testet är för när katalas är frånvarande, men det kunde ha varit katalas närvarande. De motstridiga resultaten av metagenome binning och katalastestet påverkade detta misstag. Jag tror baserat på all denna information, som min mikrob är i själva verket micrococcus luteus som föreslagits av PATRIC metagenome binning test, och kronan (Figur 1).
Sammanfattningsvis var några av mina resultat ofullständiga och motstridiga., Detta är sannolikt antingen en orsak till mänskligt fel, unpure kulturer, eller inte använda agar plattor som är tillräckligt färska för testet. Jag tror att denna kultur var mestadels Micrococcus luteus baserat på Kaiju och metagenome binning resultat. Syreklassen och mikrobens gram-positivitet matchar också med Mikrokockus luteus. I framtiden arbetar med denna mikrobe, skulle jag förmodligen vilja rena kulturen mer och göra om testen.
Bassis CM, AL Tang, VB Unga, och MA Pynnonen (2014). Mikrobiota i näshålan hos friska vuxna. Mikrobiomet 2(27).,
Rawis M, AK och Ellis (2019). Mikrobiomet i näsan. Annaler av Allergi, Astma och immunologi 122 (1): 17-24.
(2010) Vad är mikrober? Institutet för kvalitet och effektivitet inom hälso-och sjukvård.