Introdução:
os Micróbios são demasiado pequeno para ser visto a olho nu; eles podem sobreviver em condições que muitos pensam que são inabitável, como o ambiente anaeróbio no rúmen de vacas, nascentes de água quente e fria, águas Antárticas (o Que são micróbios, 2010). Milhões de micróbios vivem dentro e no corpo humano e ambos podem nos ajudar a sobreviver ou nos deixar doentes, menos de 1% das bactérias causam doenças (o que são micróbios, 2010).,
o microbioma da cavidade nasal consiste principalmente nos filos Actinobacteria, Firmicutes e Proteobacteria (Bassis et al. 2014). O microbioma da cavidade nasal também pode mudar em resposta a fatores ambientais como localização geográfica e Higiene (Rawis et al. 2019).Micrococcus luteus é encontrado em muitos lugares, incluindo pele, solo, poeira, água, ar, boca, mucosas, orofaringe e trato respiratório superior do ser humano (Wikipedia, Micrococcus luteus, 2019). É um gram positivo, em forma de coccus, e contém catalase. Este micróbio forma colónias grandes e redondas., It can be easily be confunded for staphylococci, as they are very similar morphologically and physiologically (Wikipedia, Staphlyococcus Aureus).o meu objectivo nesta experiência era isolar, caracterizar e identificar uma colónia bacteriana que surgiu de uma amostra tirada do nariz do meu colega de quarto. Eu hipotetized que seria uma bactéria comumente encontrada nas cavidades nasais e provavelmente a partir do phyla acima mencionado, Assim que provavelmente faria melhor viver em um ambiente aeróbico, úmido, e quente.
métodos:
escolhi experimentar bactérias do interior do nariz do meu colega de quarto., Para a amostra, usei cotonetes estéreis e raspei-os em placas TSA. Mantive a placa à temperatura ambiente durante 7 dias, e depois seleccionei uma colónia para purificar usando o método de pura cultura. Repeti este processo mais três vezes para purificar ainda mais a colónia. Uma vez que a cultura foi considerada pura o suficiente, inoculei um tubo de inclinação.
I performed many tests to find out the colony morphology and physiology., A fim de determinar as características fisiológicas da cultura, como forma celular, arranjo, e se era gram positivo ou negativo, o que ajuda a determinar o tipo de parede celular do micróbio, eu realizei uma mancha de grama. Usei uma tira de ensaio de oxidase e água para determinar se o citocromo C oxidase estava presente, e realizei um teste de catalase para determinar se a catalase estava presente. Também fiz um teste de tioglicolato líquido para determinar a classe de oxigénio das bactérias., Cultivei a minha bactéria numa placa azul de metileno (EMB) de eosina para ver se podia fermentá-la e se podia crescer com azul de metileno que selecciona bactérias gram-negativas. Também o cultivei numa placa de MacConkey (MAC) para ver se podia fermentar lactose e se podia crescer com ambos os sais de violeta cristalina e bílis para confirmar se era gram negativo ou positivo. Usei um teste de estreptococos API para determinar mais açúcares que as bactérias podem fermentar.cultivei o meu caldo de soja Triptico durante uma semana para preparar a extracção do ADN., Extraí o ADN usando o Kit de adn do “PowerSoil” (fabricado pela Qiagen) de acordo com as instruções do fabricante. A amostra foi então sequenciada usando a tecnologia Illumina MiSeq no laboratório de DNA da UAF. Usei o software PATRIC para executar um binning metagenome e atribuir uma taxonomia às bactérias.resultados: a colónia demorou 16 dias para ser purificada. A coloração gram deste micróbio mostrou que é gram positivo porque manchou roxo. Este micróbio é em forma de coccus e forma-se em tetrads. A colónia forma-se como uma bolha amarela e redonda brilhante., Os testes da catalase e da oxidase deram negativo, porque o teste da catalase não formou bolhas, e o teste da oxidase não viu uma mudança de cor. Os testes da oxidase para verificar se o micróbio contém citocromo c oxidase. O teste da catalase para ver se o micróbio contém catalase. O teste de tioglicalato de fluido mostrou que a bactéria era um aeróbio obrigatório porque o crescimento estava concentrado no topo do tubo na região rosa. A ágar MacConkey mostrou muito pouco crescimento, e não teve uma mudança de cor, indicando que o micróbio era gram positivo e não um fermentador., O ágar EMB não mostrou crescimento ou mudança de cor, também indicando que o micróbio era gram positivo e um não-fermentador.
a atribuição taxonómica deste micróbio foi micrococcus luteus porque foi o único bin que PATRIC deu. Tinha 27.372 contigs em montagem. Tem múltiplos genes de Resistência a antibióticos, incluindo dihidropteroato sintase, diéster fosfodiesterase glicerofosforilo e proteínas ribossomais SSU.
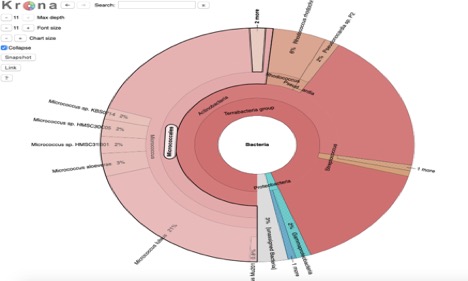 Figura 1. Krona chart of microbe shows bacterial classes thought to be present in the sample.
Figura 1. Krona chart of microbe shows bacterial classes thought to be present in the sample.
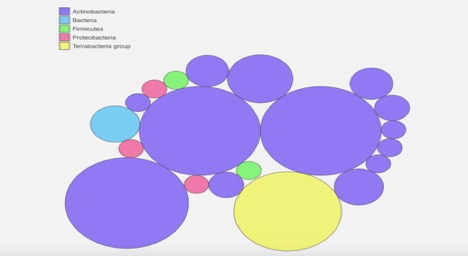 Figura 2., Kaiju webserver metagenome binning analysis chart. Mostra que a amostra contém bactérias do grupo Terrabacteria. É principalmente Actinobacteria, mas algumas proteobactérias e Firmículas também estão na amostra.o binning metagenoma de kaiju mostra que a amostra de micróbios não é completamente pura (Figura 2). Mostra que é principalmente Actinobacteria, com algumas firmículas, e proteobactérias misturadas (Figura 2). Isto corresponde ao binning METAGENOME PATRIC que também mostrou algumas impurezas (Figura 1).,
Figura 2., Kaiju webserver metagenome binning analysis chart. Mostra que a amostra contém bactérias do grupo Terrabacteria. É principalmente Actinobacteria, mas algumas proteobactérias e Firmículas também estão na amostra.o binning metagenoma de kaiju mostra que a amostra de micróbios não é completamente pura (Figura 2). Mostra que é principalmente Actinobacteria, com algumas firmículas, e proteobactérias misturadas (Figura 2). Isto corresponde ao binning METAGENOME PATRIC que também mostrou algumas impurezas (Figura 1).,
discussão:
Como o micróbio é gram positivo, isto significa que tem uma grande camada de peptidoglicano e carece de uma camada de lipopolissacarídeo. A ágar MacConkey é seletiva para o gram-negativo e é por isso que o meu micróbio não mostrou muito crescimento nele, e porque não mudou de cores significa que não fermentou a lactose. A placa de EMB também é seletiva para bactérias gram-negativo, e é provavelmente por isso que a bactéria não cresceu nela. A classe de oxigênio do micróbio, aeróbio obrigatório, coincide com as previsões que eu tinha feito sobre isso, porque a bactéria foi originalmente originada em uma narina., Wikipedia also says that Micrococcus luteus is an obligate aerobe, backing up what my results show (2019).
os resultados do teste da oxidase sugerem que o micróbio não contém oxidase, apesar do que o teste de aglutinação metagenómica mostrou. O teste da catalase também indicou que o micróbio não tem catalase, apesar do teste de binning metagenômico sugerindo isso. Estas discrepâncias podem ser devido ao erro humano, à cultura impura, ou a uma velha placa de ágar., A falta de resultados das tiras de teste da API sugere que o I usou a tira de teste errada, eu provavelmente precisava usar o teste de Staph em vez do teste de Strep, porque o teste de Strep é para quando Catalase está ausente, mas poderia ter havido catalase presente. Os resultados conflitantes do metagenome binning e do teste catalase influenciaram este erro. Penso que, com base em toda esta informação, o meu micróbio é, de facto, o micrococcus luteus, tal como sugerido pelo teste PATRIC metagenome binning, e pela coroa (Figura 1).em conclusão, alguns dos meus resultados foram inconclusivos e conflituosos., Isto é provavelmente uma causa de erro humano, culturas impuras, ou não usando placas de ágar que são frescas o suficiente para o teste. Eu acho que esta cultura era principalmente Micrococcus luteus baseado nos resultados Kaiju e metagenome binning. A classe de oxigênio e a positividade gram do micróbio também coincidem com a do Micrococcus luteus. No futuro trabalha com este micróbio, eu provavelmente gostaria de purificar mais a cultura e refazer os testes.Bassis CM, AL Tang, VB Young, e MA Pynnonen (2014). A microbiota nasal de adultos saudáveis. Microbiome 2 (27).,Rawis M e Ak Ellis (2019). O microbioma do nariz. Anais de Alergia, Asma e Imunologia 122 (1): 17-24.o que são micróbios? Instituto de qualidade e eficiência nos cuidados de saúde.