wprowadzenie:
drobnoustroje są zbyt małe, aby można je było zobaczyć gołym okiem; mogą przetrwać w warunkach, które wielu uważałoby za niemożliwe, takich jak środowisko beztlenowe w żwaczu krów, gorących źródłach i zimnych wodach Antarktyki (What are microbes, 2010). Miliony drobnoustrojów żyją zarówno na iw ludzkim ciele i mogą pomóc nam przetrwać lub sprawić, że zachorujemy, mniej niż 1% bakterii powoduje choroby (What are microbes, 2010).,
mikrobiom jamy nosowej składa się głównie z phyla Actinobacteria, Firmicutes i Proteobacteria (Bassis et al. 2014). Mikrobiom jamy nosowej może również ulec zmianie w odpowiedzi na czynniki środowiskowe, takie jak lokalizacja geograficzna i higiena (ravis et al. 2019).
Micrococcus luteus występuje w wielu miejscach, w tym w skórze, glebie, kurzu, wodzie, powietrzu, ustach, śluzach, gardle i górnych drogach oddechowych ludzi (Wikipedia, Micrococcus luteus, 2019). Jest gram-dodatnim mikrobem w kształcie kokonu i zawiera katalazę. Mikrob ten tworzy duże, okrągłe kolonie., Można go łatwo pomylić z gronkowcami, ponieważ są bardzo podobne morfologicznie i fizjologicznie (Wikipedia, Staphylococcus Aureus).
moim celem w tym eksperymencie było wyizolowanie, scharakteryzowanie i zidentyfikowanie Kolonii bakteryjnej, która powstała na podstawie próbki pobranej z nosa mojej współlokatorki. Postawiłem hipotezę, że będzie to bakteria powszechnie występująca w jamach nosowych i prawdopodobnie od wspomnianej phyla, więc prawdopodobnie najlepiej będzie żyć w środowisku tlenowym, wilgotnym i ciepłym.
metody:
wybrałem pobranie bakterii z wnętrza nosa mojej współlokatorki., Do próbkowania użyłam sterylnych wacików bawełnianych i posmarowałam je płytkami TSA. Trzymałem płytkę w temperaturze pokojowej przez 7 dni, a następnie wybrałem kolonię do oczyszczenia za pomocą metody płytkowej Pure culture streak. Powtórzyłem ten proces jeszcze trzy razy, aby jeszcze bardziej oczyścić kolonię. Kiedy kultura została uznana za wystarczająco czystą, zaszczepiłem skośną rurkę.
wykonałem wiele badań, aby poznać morfologię i fizjologię Kolonii., W celu określenia fizjologicznych cech kultury, takich jak kształt, rozmieszczenie komórek i czy jest to gram dodatnia czy ujemna, co pomaga określić typ ściany komórkowej drobnoustrojów, wykonałem plamę grama. Użyłem paska testowego oksydazy i wody, aby określić, czy oksydaza cytochromu C była obecna, i wykonałem test catalazy, aby określić, czy catalaza była obecna. Zrobiłem też test tioglikolanu w płynie, aby określić klasę tlenu bakterii., Hodowałem moje bakterie na płycie eozyny błękitu metylenowego (EMB), aby sprawdzić, czy może fermentować laktozę i czy może rosnąć z błękitem metylenowym, który wybiera bakterie gram-ujemne. Wyhodowałem go również w płytce MacConkey (MAC), aby sprawdzić, czy może fermentować laktozę i czy może rosnąć zarówno z fioletem krystalicznym, jak i solami żółciowymi, aby dodatkowo potwierdzić, czy był gram-ujemny lub dodatni. Użyłem testu API paciorkowca, aby określić więcej cukrów, które bakterie mogą fermentować.
wyhodowałem swój izolowany w Tryptycznym bulionie sojowym (TSB) przez tydzień, aby przygotować się do ekstrakcji DNA., DNA wyodrębniłem za pomocą zestawu POWERSOIL DNA (produkowanego przez Qiagen) zgodnie z instrukcjami producenta. Następnie próbka została zsekwencjonowana przy użyciu technologii Illumina MiSeq w laboratorium DNA Core Lab UAF. Użyłem oprogramowania PATRIC do wykonania wiązania metagenomu i przypisania taksonomii bakteriom.
wyniki:
oczyszczanie Kolonii trwało 16 dni. Plama grama tego drobnoustroju wykazała, że jest gram dodatnia, ponieważ zabarwiła się na fioletowo. Drobnoustrój ten ma kształt kokonu i tworzy tetrady. Kolonia tworzy żółtą, błyszczącą okrągłą plamę., Testy catalazy i oksydazy wyszły negatywnie, ponieważ test catalazy nie tworzył pęcherzyków, a test oksydazy nie widział zmiany koloru. Test oksydazy sprawdza, czy drobnoustrój zawiera oksydazę cytochromu C. Test catalazy sprawdza, czy mikrob zawiera katalazę. Badanie tioglikolanu w płynie wykazało, że bakteria była zobowiązana do aerobiku, ponieważ wzrost był skoncentrowany w górnej części rurki w regionie różowym. Agar Macconkeya wykazywał bardzo niewielki wzrost i nie miał zmiany koloru, co wskazuje, że drobnoustrój był gram dodatni, a nie fermentator., Agar EMB nie wykazywał wzrostu ani zmiany koloru, wskazując również, że drobnoustrój był gram dodatni i nie fermentował.
taksonomicznym przypisaniem tego drobnoustroju był micrococcus luteus, ponieważ był to jedyny gatunek, który podał PATRIC. 27 372 mieszkańców. Ma wiele genów oporności na antybiotyki, w tym syntazę dihydropteronianową, fosfodiesterazę glicerofosforylową i białka rybosomalne SSU.
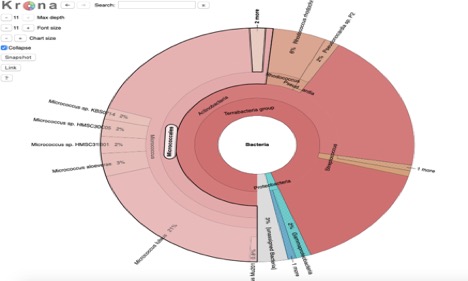 Rysunek 1. Wykres Krona mikrob pokazuje klasy bakterii, które uważa się za obecne w próbce.
Rysunek 1. Wykres Krona mikrob pokazuje klasy bakterii, które uważa się za obecne w próbce.
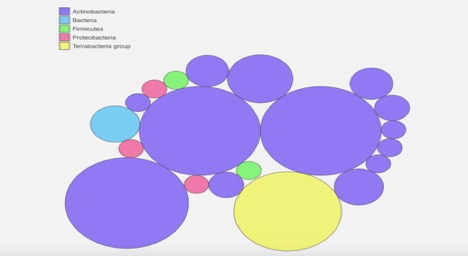 Rysunek 2., Kaiju webserver metagenome binning analysis chart. Pokazuje, że próbka zawiera bakterie z grupy Terrabakterii. Jest to głównie Actinobacteria, ale niektóre Proteobacteria i Firmicules są w próbce, jak również.
Rysunek 2., Kaiju webserver metagenome binning analysis chart. Pokazuje, że próbka zawiera bakterie z grupy Terrabakterii. Jest to głównie Actinobacteria, ale niektóre Proteobacteria i Firmicules są w próbce, jak również.
Wiązanie metagenomu kaiju pokazuje, że próbka drobnoustrojów nie jest całkowicie czysta (Rysunek 2). To pokazuje, że jest to głównie Actinobacteria, z niektórych firmicules, i proteobacteria mieszane w (Rysunek 2). To pasuje do PATRIC metagenome binning, które również wykazały pewne zanieczyszczenia (Rysunek 1).,
dyskusja:
ponieważ drobnoustrój jest gram dodatni, oznacza to, że ma dużą warstwę peptydoglikanu i nie ma warstwy lipopolisacharydowej. Agar Macconkeya jest selektywny dla gram-ujemnych, dlatego mój drobnoustrój nie wykazywał na nim dużego wzrostu, a ponieważ nie zmienił kolorów, oznacza to, że nie fermentował laktozy. Płyta EMB jest również selektywna dla bakterii gram-ujemnych, co prawdopodobnie dlatego bakterie nie rosły na niej. Klasa tlenowa drobnoustrojów, obligate aerobe, zgadza się z przewidywaniami, które na ten temat poczyniłem, ponieważ bakterie były pierwotnie pozyskiwane w nozdrzach., Wikipedia mówi również, że Micrococcus luteus jest aerobikiem obowiązkowym, potwierdzając to, co pokazują moje wyniki(2019).
wyniki testu oksydazy sugerują, że drobnoustrój nie zawiera oksydazy, pomimo tego, co wykazał test wiązania metagenomu. Badanie catalazy wykazało również, że drobnoustrój nie ma katalazy, pomimo sugerującego go testu wiązania metagenomicznego. Różnice te mogą być spowodowane błędem ludzkim, nieuregulowaną kulturą lub starą płytą agarową., Brak wyników pasków testowych API sugeruje, że użyłem niewłaściwego paska testowego, prawdopodobnie potrzebowałem użyć testu gronkowca zamiast testu paciorkowca, ponieważ test paciorkowca jest dla katalazy nieobecnej, ale mogła być obecna catalaza. Sprzeczne wyniki wiązania metagenomu i testu katalazy wpłynęły na ten błąd. Myślę, że na podstawie tych wszystkich informacji, że mój mikrob jest w rzeczywistości micrococcus luteus, jak sugeruje test wiązania metagenomu Patrica i korona (Rysunek 1).
podsumowując, niektóre z moich wyników były niejednoznaczne i sprzeczne., Jest to prawdopodobnie albo przyczyną błędu ludzkiego, nieure kultur, lub nie za pomocą płyt agarowych, które są wystarczająco świeże do badania. Myślę, że kultura ta była głównie Micrococcus luteus w oparciu o wyniki wiązania Kaiju i metagenomu. Klasa tlenu i gram dodatni mikrob również pasuje do Micrococcus luteus. W przyszłości pracuje z tym mikrobem, prawdopodobnie chciałbym bardziej oczyścić kulturę i powtórzyć testy.
Bassis CM, AL Tang, VB Young, And MA Pynnonen (2014). Mikrobiota jamy nosowej zdrowych dorosłych. Mikrobiom 2(27),
Rawis M, i AK Ellis (2019). Mikrobiom nosa. Annals of Allergy, Asthma and Immunology 122(1):17-24.
(2010) czym są mikroby? Instytut jakości i efektywności w Ochronie Zdrowia.