Innledning:
Mikrober er for lite til å kunne sees med det blotte øyet; de kan overleve under forhold som mange vil synes er unlivable som den anaerobe miljø i vommen av kyr, hot springs, og det kalde Antarktiske farvann (Hva er mikrober, 2010). Millioner av mikrober live både på og i kroppen og kan både gjøre hjelpe oss til å overleve eller gjøre oss syke, mindre enn 1% av bakterier forårsake sykdom (Hva er mikrober, 2010).,
nesehulen microbiome først og fremst består av phyla Actinobacteria, Firmicutes og Proteobacteria (Bassis et al. 2014). Den microbiome av nesehulen kan også endre i respons til miljømessige faktorer som geografisk plassering, og hygiene (Rawis et al. 2019).
Micrococcus luteus er funnet i mange steder, inkludert huden, jord, støv, vann, luft, munn, mucosae, orofarynx, og øvre luftveier hos mennesker (Wikipedia, Micrococcus luteus, 2019). Det er en gram-positiv, coccus formet mikrobe, og inneholder catalase. Dette mikrobe danner store, runde kolonier., Det kan være lett forveksles med stafylokokker, som de er svært lik morphologically og fysiologisk (Wikipedia, Staphlyococcus Aureus).
målet Mitt i dette eksperimentet var å isolere, karakterisere og identifisere en bakteriell koloni som oppsto fra en prøve tatt fra min romkamerat nese. Jeg hypotese om at det ville være en bakterie som vanligvis finnes i nese hulrom og sannsynligvis fra de nevnte phyla, så det ville sannsynligvis gjøre det beste som bor i en aerobic, fuktig og varmt miljø.
Metoder:
jeg valgte å prøve bakterier fra innsiden min romkamerat nese., For eksempel, jeg brukte sterilt bomull vattpinner og stripete dem på TSA-plater. Jeg holdt plate ved romtemperatur i 7 dager, og deretter valgt en koloni for å rense bruke ren kultur strek plate metode. Jeg gjentok denne prosessen tre ganger for ytterligere å rense kolonien. Når kultur ble ansett som ren nok, jeg inokulert en skrå rør.
jeg har utført mange tester for å finne ut kolonien morfologi og fysiologi., For å finne ut fysiologiske egenskaper av kultur som cellen form, arrangement, og om det var gram-positive eller negative, som hjelper deg med å finne cellevegg type mikrobe, jeg utført en gramfarging. Jeg brukte en oksidase test strip og vann for å finne ut om cytokrom C oksidase var til stede, og utført en catalase test for å fastslå om catalase var til stede. Jeg gjorde også en væske tioglykolat test for å fastslå bakterier er oksygen klasse., Jeg vokste min bakterier på en Eosin-Methylene Blå (EMB) plate for å se om det kunne gjære laktose og om det kunne vokse med methylene blå som velger for gram-negativ bakterier. Jeg vokste også det i en MacConkey (MAC) plate for å se om det kunne gjære laktose og om det kunne vokse med både crystal violet og galle salter å ytterligere bekrefte om det var gram-negativ eller positiv. Jeg brukte en API Strep-test for å finne ut mer av sukker bakterier kunne gjære.
jeg vokste min isolert i Tryptic Soya Buljong (TSB) for en uke å forberede seg til DNA-ekstraksjon., Jeg ekstrahert DNA ved hjelp av PowerSoil DNA-kit (produsert av Qiagen), etter produsentens anvisninger. Prøven ble deretter sekvensert ved hjelp av Illumina MiSeq teknologi i UAF er DNA-Core Lab. Jeg brukte PATRIC programvare for å utføre en metagenome gruppering og til å tilordne en taksonomi for bakterier.
Resultater:
kolonien tok 16 dager for å bli renset. Den gramfarging av denne mikrobe viste at det er gram-positive fordi det farget lilla. Dette mikrobe er coccus formet og former i tetrads. Kolonien former som et gult, skinnende runde blob., Den catalase og oksidase tester kom opp negative, fordi catalase test gjorde ikke dannes bobler, og oksidase test fikk ikke se en fargeendring. Den oksidase test tester for å se om mikrobe inneholder cytokrom c oksidase. Den catalase test tester for å se om mikrobe inneholder catalase. Væsken thioglycallate test viste at bakterier var en forplikte aerobe fordi veksten var konsentrert på toppen av røret i den rosa regionen. Den MacConkey-agar viste svært liten vekst, og har ikke en endring i farge, indikerer at de mikrobe var gram positive og ikke en fermenter., Den EMB agar viste ingen vekst eller endring i farge, også indikerer mikrobe var gram positive og en ikke-fermenter.
Den taksonomisk oppdrag av denne mikrobe var micrococcus luteus fordi det var den eneste utskuff som PATRIC ga. Det hadde 27,372 contigs i forsamlingen. Det har flere antibiotika resistens gener inkludert dihydropteroate syntase, glycerophosphoryl diester fosfodiesterase, og SSU ribosom-proteiner.
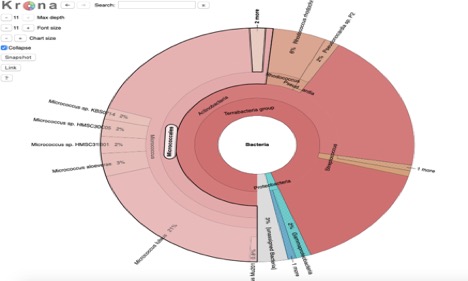 Figur 1. Kroner figur av mikrobe viser bakteriell klasser tenkt å være til stede i prøven.
Figur 1. Kroner figur av mikrobe viser bakteriell klasser tenkt å være til stede i prøven.
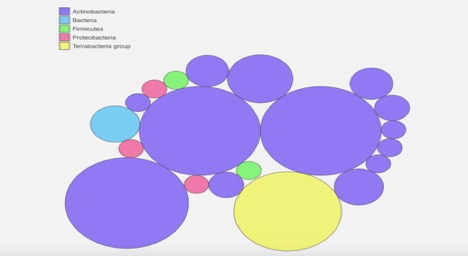 Figur 2., Kaiju webserver metagenome gruppering analyse diagram. Det viser at prøven ikke inneholder bakterier fra Terrabacteria gruppe. Det er for det meste Actinobacteria, men noen Proteobacteria og Firmicules er i utvalget som godt.
Figur 2., Kaiju webserver metagenome gruppering analyse diagram. Det viser at prøven ikke inneholder bakterier fra Terrabacteria gruppe. Det er for det meste Actinobacteria, men noen Proteobacteria og Firmicules er i utvalget som godt.
kaiju metagenome gruppering viser at mikrobe eksempel er ikke fullstendig ren (Figur 2). Det viser at det er stort sett Actinobacteria, med noen firmicules ,og proteobacteria blandet i (Figur 2). Dette samsvarer med opp med PATRIC metagenome gruppering som viste også noen urenheter (Figur 1).,
Diskusjon:
Som mikrobe er gram-positive dette betyr at det har en stor peptidoglycan lag, og mangler en lipopolysaccharide lag. Den MacConkey-agar er selektive for gram-negativ, noe som er grunnen til at mitt mikrobe ikke viser mye vekst på det, og fordi det ikke endre fargene betyr det at det ikke gjære laktose. Den EMB plate er også selektiv for gram-negative bakterier som sannsynligvis er grunnen til at bakterier ikke vokser på det. Oksygen klasse av mikrobe, forplikte aerobe, matcher opp med spådommer jeg hadde gjort om det fordi bakteriene ble opprinnelig hentet i en nesebor., Wikipedia sier også at Micrococcus luteus er en forplikte aerobe, sikkerhetskopiere hva min resultatene viser (2019).
oksidase test resultatene tyder på at mikrobe ikke inneholder oksidase, til tross for hva metagenome gruppering test viste. Den catalase test også indikert at mikrobe ikke har catalase, til tross for den metagenomic gruppering test tyder på det. Disse avvikene kan være på grunn av menneskelige feil, uvirkelig kultur, eller en gammel agar plate., API test strips » mangel på resultater antyder at jeg brukte feil test strip, jeg sannsynligvis nødvendig for å bruke Staph test i stedet for Strep-test, fordi Strep-test er for når Catalase er fraværende, men det kunne ha vært catalase til stede. De motstridende resultatene av metagenome gruppering og catalase test påvirket denne feilen. Jeg tror basert på all denne informasjonen, som min mikrobe er faktisk micrococcus luteus som foreslått av PATRIC metagenome gruppering test, og krone (Figur 1).
I konklusjonen, noen av mine resultater var mangelfulle og motstridende., Dette er sannsynligvis enten en årsak til menneskelige feil, uvirkelig kulturer, eller ikke bruker agar plater som er frisk nok til testen. Jeg tror at denne kulturen var for det meste Micrococcus luteus basert på Kaiju og metagenome gruppering resultater. Oksygen klasse og gram positivitet av mikrobe også matcher opp med Micrococcus luteus. I fremtiden jobber med dette mikrobe, ville jeg sannsynligvis vil rense kultur mer og gjenta testene.
Bassis CM, AL Tang, VB Unge, og MA Pynnonen (2014). Nesehulen microbiota av friske voksne. Microbiome 2(27).,
Rawis M, og AK Ellis (2019). Den microbiome av nesen. Annals of Allergy, Asthma, og Immunologi 122(1):17-24.
(2010) Hva er mikrober? Institutt for Kvalitet og Effektivitet i helsevesenet.