Einführung:
Mikroben sind zu klein, um gesehen werden durch die bloßen Auge, können Sie überleben in den Bedingungen, dass viele denken, würde unbewohnbar wie der anaeroben Umgebung im Pansen von Kühen, heiße Quellen, und kalte antarktische Wasser (Was sind Mikroben, 2010). Millionen von Mikroben leben sowohl auf als auch im menschlichen Körper und können uns beide helfen, zu überleben oder uns krank zu machen, weniger als 1% der Bakterien verursachen Krankheiten (Was sind Mikroben, 2010).,
Das Nasenhöhlenmikrobiom besteht primär aus den Phyla Actinobacteria, Firmicutes und Proteobacteria (Bassis et al. 2014). Das Mikrobiom der Nasenhöhle kann sich auch als Reaktion auf Umweltfaktoren wie geografische Lage und Hygiene ändern (Rawis et al. 2019).
Micrococcus luteus kommt an vielen Stellen vor, darunter Haut, Boden, Staub, Wasser, Luft, Mund, Schleimhäute, Oropharynx und obere Atemwege des Menschen (Wikipedia, Micrococcus luteus, 2019). Es ist eine grampositive, kokkusförmige Mikrobe und enthält Katalase. Diese Mikrobe bildet große, runde Kolonien., Es kann leicht mit Staphylokokken verwechselt werden, da sie morphologisch und physiologisch sehr ähnlich sind (Wikipedia, Staphylococcus Aureus).
Mein Ziel in diesem Experiment war es, eine Bakterienkolonie zu isolieren, zu charakterisieren und zu identifizieren, die aus einer Probe aus der Nase meines Mitbewohners entstanden ist. Ich stellte die Hypothese auf, dass es sich um ein Bakterium handeln würde, das häufig in den Nasenhöhlen vorkommt und wahrscheinlich aus der oben genannten Phyla stammt, sodass es wahrscheinlich am besten in einer aeroben, feuchten und warmen Umgebung leben würde.
Methoden:
Ich habe Bakterien aus der Nase meines Mitbewohners entnommen., Zur Probe habe ich sterile Wattestäbchen verwendet und sie auf TSA-Platten gestreift. Ich hielt die Platte 7 Tage lang bei Raumtemperatur und wählte dann eine Kolonie aus, um sie mit der Pure Culture Streak Plate-Methode zu reinigen. Ich wiederholte diesen Vorgang noch dreimal, um die Kolonie weiter zu reinigen. Sobald die Kultur als rein genug angesehen wurde, impfte ich eine schräge Röhre.
Ich habe viele Tests durchgeführt, um die Morphologie und Physiologie der Kolonie herauszufinden., Um physiologische Eigenschaften der Kultur wie Zellform, Anordnung und ob grampositiv oder negativ zu bestimmen, was zur Bestimmung des Zellwandtyps der Mikrobe beiträgt, führte ich einen Gramfleck durch. Ich verwendete einen Oxidase-Teststreifen und Wasser, um festzustellen, ob Cytochrom-C-Oxidase vorhanden war, und führte einen Katalasetest durch, um festzustellen, ob Katalase vorhanden war. Ich habe auch einen Flüssigkeitsthioglykolattest durchgeführt, um die Sauerstoffklasse der Bakterien zu bestimmen., Ich wuchs meine Bakterien auf einer Eosin Methylenblau (EMB) Platte, um zu sehen, ob es Laktose gären könnte und ob es mit Methylenblau wachsen könnte, die für gramnegative Bakterien auswählt. Ich habe es auch in einer MacConkey-Platte (MAC) angebaut, um zu sehen, ob es Laktose fermentieren kann und ob es sowohl mit Kristallviolett-als auch mit Gallensalzen wachsen kann, um weiter zu bestätigen, ob es gramnegativ oder positiv ist. Ich habe einen API-Strep-Test verwendet, um mehr Zucker zu bestimmen, den die Bakterien gären könnten.
Ich bin eine Woche lang in Tryptic Soy Broth (TSB) aufgewachsen, um mich auf die DNA-Extraktion vorzubereiten., Ich habe die DNA mit dem PowerSoil DNA Kit (hergestellt von Qiagen) gemäß den Anweisungen des Herstellers extrahiert. Die Probe wurde dann mit der Illumina MiSeq-Technologie im DNA Core Lab von UAF sequenziert. Ich habe die PATRIC-Software verwendet, um ein Metagenom-Binning durchzuführen und den Bakterien eine Taxonomie zuzuweisen.
Ergebnisse:
Die Reinigung der Kolonie dauerte 16 Tage. Der Gram-Fleck dieser Mikrobe zeigte, dass er grampositiv ist, weil er lila gefärbt ist. Diese Mikrobe ist kokkusförmig und bildet sich in Tetraden. Die Kolonie bildet sich als gelber, glänzender runder Fleck., Die Katalase – und die Oxidase-Tests waren negativ, da der Katalase-Test keine Blasen bildete und der Oxidase-Test keine Farbänderung sah. Der Oxidase-Test prüft, ob die Mikrobe Cytochrom-c-Oxidase enthält. Der Katalase-Test prüft, ob die Mikrobe Katalase enthält. Der Flüssigkeitsthioglycallattest zeigte, dass die Bakterien eine obligate Aerobe waren, da das Wachstum an der Oberseite der Röhre im rosa Bereich konzentriert war. Der MacConkey-Agar zeigte sehr wenig Wachstum und hatte keine Farbveränderung, was darauf hindeutet, dass die Mikrobe grampositiv und kein Fermenter war., Der EMB-Agar zeigte kein Wachstum oder keine Veränderung der Farbe, was auch darauf hindeutet, dass die Mikrobe grampositiv und ein Nicht-Fermenter war.
Die taxonomische Zuordnung dieser Mikrobe war Micrococcus luteus, weil es der einzige Bin war, den PATRIC gab. Es hatte 27,372 contigs in der Montage. Es hat mehrere Antibiotikaresistenzgene, einschließlich Dihydropteroatsynthase, Glycerophosphoryldiesterphosphodiesterase und SSU-ribosomale Proteine.
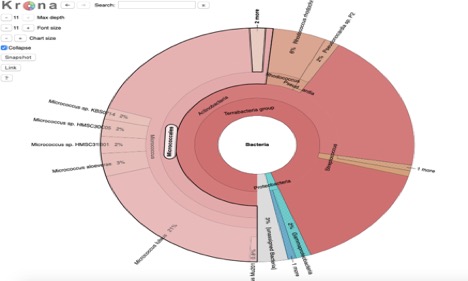 Abbildung 1. Krona-Diagramm der Mikrobe zeigt Bakterienklassen, von denen angenommen wird, dass sie in der Probe vorhanden sind.
Abbildung 1. Krona-Diagramm der Mikrobe zeigt Bakterienklassen, von denen angenommen wird, dass sie in der Probe vorhanden sind.
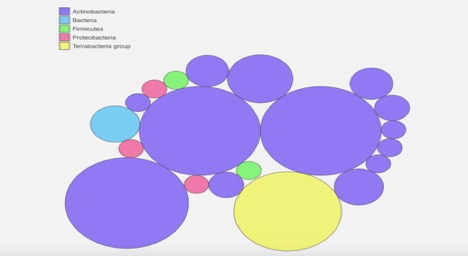 Abbildung 2., Kaiju webserver Metagenom-binning-Analyse-Diagramm. Es zeigt, dass die Probe Bakterien aus der Terrabakteriegruppe enthält. Es handelt sich hauptsächlich um Aktinobakterien, aber auch einige Proteobakterien und Firmicules befinden sich in der Probe.
Abbildung 2., Kaiju webserver Metagenom-binning-Analyse-Diagramm. Es zeigt, dass die Probe Bakterien aus der Terrabakteriegruppe enthält. Es handelt sich hauptsächlich um Aktinobakterien, aber auch einige Proteobakterien und Firmicules befinden sich in der Probe.
Das kaiju Metagenom binning zeigt, dass die Mikrobenprobe nicht vollständig rein ist (Abbildung 2). Es zeigt, dass es sich hauptsächlich um Aktinobakterien mit einigen Firmicules und eingemischten Proteobakterien handelt (Abbildung 2). Dies stimmt mit dem PATRIC Metagenom Binning überein, das ebenfalls einige Verunreinigungen zeigte (Abbildung 1).,
Diskussion:
Da die Mikrobe grampositiv ist, bedeutet dies, dass sie eine große Peptidoglycanschicht und keine Lipopolysaccharidschicht aufweist. Der MacConkey-Agar ist selektiv für gramnegativ, weshalb meine Mikrobe nicht viel Wachstum zeigte und weil sie ihre Farben nicht änderte, bedeutet dies, dass sie die Laktose nicht fermentierte. Die EMB-Platte ist auch selektiv für gramnegative Bakterien, weshalb die Bakterien wahrscheinlich nicht darauf gewachsen sind. Die Sauerstoffklasse der Mikrobe, obligate Aerobe, stimmt mit den Vorhersagen überein, die ich darüber gemacht hatte, weil die Bakterien ursprünglich in einem Nasenloch stammen., Wikipedia sagt auch, dass Micrococcus luteus ein obligater Aerobe ist, was meine Ergebnisse zeigen (2019).
Die Oxidase-Testergebnisse legen nahe, dass die Mikrobe keine Oxidase enthält, obwohl der Metagenom-Binning-Test gezeigt hat. Der Katalase-Test zeigte auch, dass die Mikrobe keine Katalase hat, obwohl der metagenomische Binning-Test dies nahelegt. Diese Diskrepanzen können auf menschliches Versagen, unreine Kultur oder eine alte Agarplatte zurückzuführen sein., Der Mangel an Ergebnissen der API-Teststreifen legt nahe, dass ich, wenn ich den falschen Teststreifen verwendet habe, wahrscheinlich den Staph-Test anstelle des Strep-Tests verwenden musste, da der Strep-Test für den Fall gilt, dass Katalase fehlt, aber Katalase vorhanden sein könnte. Die widersprüchlichen Ergebnisse des Metagenom-Binnings und des Katalase-Tests beeinflussten diesen Fehler. Ich denke, basierend auf all diesen Informationen, dass meine Mikrobe tatsächlich Micrococcus luteus ist, wie vom PATRIC metagenome binning Test und der Krone vorgeschlagen (Abbildung 1).
Abschließend waren einige meiner Ergebnisse nicht schlüssig und widersprüchlich., Dies ist wahrscheinlich entweder eine Ursache für menschliches Versagen, unreine Kulturen oder die Verwendung von Agarplatten, die für den Test frisch genug sind. Ich denke, dass diese Kultur hauptsächlich Micrococcus luteus war, basierend auf den Ergebnissen von Kaiju und Metagenom binning. Die Sauerstoffklasse und die Grampositivität der Mikrobe stimmen auch mit der von Micrococcus luteus überein. In zukünftigen Arbeiten mit dieser Mikrobe möchte ich wahrscheinlich die Kultur mehr reinigen und die Tests wiederholen.
Bassis CM, AL Tang, VB Junge, und Pynnonen MA (2014). Die Nasenhöhle Mikrobiota von gesunden Erwachsenen. Mikrobiom-2(27).,
Rawis M und AK Ellis (2019). Das mikrobiom der Nase. Annalen der Allergie, Asthma und Immunologie 122(1):17-24.
(2010) Was sind Mikroben? Institut für Qualität und Effizienz im Gesundheitswesen.