Introduction:
Les microbes sont trop petits pour être vus à l’œil nu; ils peuvent survivre dans des conditions que beaucoup penseraient invivables comme l’environnement anaérobie dans le rumen des vaches, les sources chaudes et les eaux froides de l’Antarctique (What are microbes, 2010). Des millions de microbes vivent à la fois sur et dans le corps humain et peuvent à la fois nous aider à survivre ou nous rendre malades, moins de 1% des bactéries causent des maladies (Quels sont les microbes, 2010).,
Le microbiome de la cavité nasale se compose principalement des phyla Actinobactéries, des Firmicutes et des protéobactéries (Bassis et al. 2014). Le microbiome de la cavité nasale peut également changer en réponse à des facteurs environnementaux tels que l’emplacement géographique et l’hygiène (Rawis et al. 2019).
Micrococcus luteus se trouve dans de nombreux endroits, y compris la peau, le sol, la poussière, l’eau, l’air, la bouche, les muqueuses, l’oropharynx et les voies respiratoires supérieures des humains (Wikipedia, Micrococcus luteus, 2019). C’est un microbe gram positif, en forme de coccus, et contient de la catalase. Ce microbe forme de grandes colonies rondes., Il peut être facilement confondu avec les staphylocoques, car ils sont très similaires morphologiquement et physiologiquement (Wikipedia, Staphlyococcus Aureus).
Mon objectif dans cette expérience était d’isoler, de caractériser et d’identifier une colonie bactérienne issue d’un échantillon prélevé sur le nez de mon colocataire. J’ai émis l’hypothèse qu’il s’agirait d’une bactérie communément trouvée dans les cavités nasales et probablement issue du phyla susmentionné, de sorte qu’elle ferait probablement mieux de vivre dans un environnement aérobie, humide et chaud.
Méthodes:
J’ai choisi de prélever des bactéries dans le nez de mon colocataire., Pour échantillonner, j’ai utilisé des cotons-tiges stériles et les ai striés sur des plaques TSA. J’ai gardé la plaque à température ambiante pendant 7 jours, puis j’ai sélectionné une colonie à purifier en utilisant la méthode pure culture streak plate. J’ai répété ce processus trois fois de plus pour purifier davantage la colonie. Une fois que la culture a été jugée assez pure, j’ai inoculé un tube oblique.
J’ai effectué de nombreux tests pour connaître la morphologie et la physiologie de la colonie., Afin de déterminer les caractéristiques physiologiques de la culture telles que la forme de la cellule, l’arrangement, et si elle était gram positif ou négatif, ce qui aide à déterminer le type de paroi cellulaire du microbe, j’ai effectué une tache de gram. J’ai utilisé une bandelette de test d’oxydase et de l’eau pour déterminer si la cytochrome C oxydase était présente, et j’ai effectué un test de catalase pour déterminer si la catalase était présente. J’ai également fait un test de thioglycolate liquide pour déterminer la classe d’oxygène de la bactérie., J’ai cultivé mes bactéries sur une plaque d’éosine de bleu de méthylène (EMB) pour voir si elle pouvait fermenter le lactose et si elle pouvait pousser avec du bleu de méthylène qui sélectionne pour les bactéries gram négatives. Je l’ai également cultivé dans une plaque MacConkey (MAC) pour voir s’il pouvait fermenter le lactose et s’il pouvait pousser avec du violet cristallin et des sels biliaires pour confirmer davantage s’il était gram négatif ou positif. J’ai utilisé un test streptococcique API pour déterminer plus de sucres que les bactéries pourraient fermenter.
J’ai cultivé mon isolé dans du bouillon de soja Tryptique (TSB) pendant une semaine pour préparer l’extraction de l’ADN., J’ai extrait l’ADN en utilisant le kit d’ADN PowerSoil (fabriqué par Qiagen) en suivant les instructions du fabricant. L’échantillon a ensuite été séquencé à l’aide de la technologie Illumina MiSeq dans le laboratoire DNA Core de l’UAF. J’ai utilisé le logiciel PATRIC pour effectuer un binning de métagénomes et pour attribuer une taxonomie aux bactéries.
Résultats:
La colonie a mis 16 jours à être purifiée. La tache de gram de ce microbe a montré qu’il est gram positif parce qu’il est taché de violet. Ce microbe est en forme de coccus et se forme en tétrades. La colonie forme une goutte ronde jaune et brillante., Les tests de catalase et d’oxydase se sont révélés négatifs, car le test de catalase n’a pas formé de bulles et le test d’oxydase n’a pas vu de changement de couleur. Le test d’oxydase teste si le microbe contient de la cytochrome c oxydase. Le test de catalase teste si le microbe contient de la catalase. Le test de thioglycallate fluide a montré que la bactérie était un aérobe obligatoire car la croissance était concentrée au sommet du tube dans la région rose. La gélose MacConkey a montré très peu de croissance et n’a pas changé de couleur, ce qui indique que le microbe était gram positif et non un fermenteur., La gélose EMB n’a montré aucune croissance ou changement de couleur, indiquant également que le microbe était gram positif et non fermenteur.
L’affectation taxonomique de ce microbe était micrococcus luteus parce que c’était le seul bac que PATRIC a donné. Il avait 27 372 contigs en assemblage. Il possède de multiples gènes de résistance aux antibiotiques, notamment la dihydroptéroate synthase, la glycérophosphoryl diester phosphodiestérase et les protéines ribosomiques SSU.
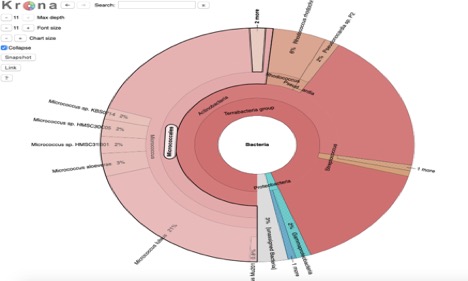 Figure 1. Le diagramme de Krona des microbes montre les classes bactériennes considérées comme présentes dans l’échantillon.
Figure 1. Le diagramme de Krona des microbes montre les classes bactériennes considérées comme présentes dans l’échantillon.
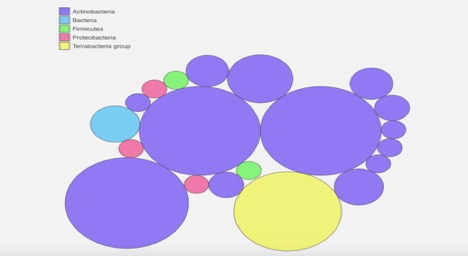 Figure 2., Kaiju serveur web métagénome binning l’analyse graphique. Il montre que l’échantillon contient des bactéries du groupe Terrabactéries. Il s’agit principalement d’actinobactéries, mais certaines protéobactéries et firmicules sont également dans l’échantillon.
Figure 2., Kaiju serveur web métagénome binning l’analyse graphique. Il montre que l’échantillon contient des bactéries du groupe Terrabactéries. Il s’agit principalement d’actinobactéries, mais certaines protéobactéries et firmicules sont également dans l’échantillon.
Le binning du métagénome kaiju montre que l’échantillon de microbe n’est pas complètement pur (Figure 2). Il montre qu’il s’agit principalement d’actinobactéries, avec quelques firmicules ,et de protéobactéries mélangées (Figure 2). Cela correspond au binning PATRIC métagénome qui a également montré quelques impuretés (Figure 1).,
Discussion:
Comme le microbe est gram positif, cela signifie qu’il a une grande couche de peptidoglycane et qu’il manque une couche de lipopolysaccharide. La gélose MacConkey est sélective pour le gram négatif, c’est pourquoi mon microbe n’a pas montré beaucoup de croissance, et parce qu’il n’a pas changé de couleur, cela signifie qu’il n’a pas fermenté le lactose. La plaque EMB est également sélective pour les bactéries à gram négatif, ce qui explique probablement pourquoi les bactéries ne se sont pas développées dessus. La classe d’oxygène du microbe, aérobe obligatoire, correspond aux prédictions que j’avais faites à ce sujet parce que la bactérie provenait à l’origine d’une narine., Wikipedia dit également que Micrococcus luteus est un aérobe obligatoire, sauvegardant ce que mes résultats montrent (2019).
Les résultats du test d’oxydase suggèrent que le microbe ne contient pas d’oxydase, malgré ce que le test de binning du métagénome a montré. Le test de catalase a également indiqué que le microbe n’a pas de catalase, malgré le test de binning métagénomique le suggérant. Ces écarts pourraient être dus à une erreur humaine, à une culture non pure ou à une vieille plaque de gélose., Le manque de résultats des bandelettes de test API suggère que j’ai utilisé la mauvaise bandelette de test, j’ai probablement dû utiliser le test staphylococcique au lieu du test streptococcique, car le test streptococcique est utilisé lorsque la catalase est absente, mais il aurait pu y avoir de la catalase présente. Les résultats contradictoires du binning de métagénome et du test de catalase ont influencé cette erreur. Je pense sur la base de toutes ces informations, que mon microbe est en fait micrococcus luteus comme suggéré par le test PATRIC metagenome binning, et la couronne (Figure 1).
En conclusion, certains de mes résultats n’étaient pas concluants et contradictoires., Ceci est probablement une cause d’erreur humaine, de cultures non pures ou de ne pas utiliser de plaques d’agar suffisamment fraîches pour le test. Je pense que cette culture était principalement Micrococcus luteus basée sur les résultats de Kaiju et de métagénome binning. La classe d’oxygène et la gram positivité du microbe correspondent également à celle de Micrococcus luteus. Dans les travaux futurs avec ce microbe, je voudrais probablement purifier davantage la culture et refaire les tests.
Bassis CM, AL Tang, VB Young et MA Pynnonen (2014). Le microbiote de la cavité nasale des adultes en bonne santé. Microbiome 2 (27).,
Rawis M, et AK Ellis (2019). Le microbiome du nez. Annales de l’Allergie, de l’Asthme et de l’Immunologie 122(1):17-24.
(2010) Que sont les microbes? Institut pour la Qualité et l’efficacité des soins de Santé.