Indledning:
Mikrober er for små til at kunne ses med det blotte øje, og de kan overleve i det forhold, at mange ville synes, er unlivable som den anaerobe miljø i vommen af køer, hot springs, og kolde Antarktiske farvande (Hvad er mikrober, 2010). Millioner af mikrober lever både på og i menneskekroppen og kan begge hjælpe os med at overleve eller gøre os syge, mindre end 1% af bakterierne forårsager sygdom (Hvad er mikrober, 2010).,
mikrobiomet i næsehulen består primært af phyla Actinobacteria, Firmicutes og Proteobacteria (Bassis et al. 2014). Mikrobiomet i næsehulen kan også ændre sig som reaktion på miljøfaktorer såsom geografisk placering og hygiejne (Ra .is et al. 2019).
Micrococcus luteus findes mange steder, herunder hud, jord, støv, vand, luft, mund, slimhinder, oropharyn.og øvre luftveje hos mennesker (Microcikipedia, Micrococcus luteus, 2019). Den er en grampositiv, coccusformet mikrobe og indeholder katalase. Denne mikrobe danner store, runde kolonier., Det kan let forveksles med stafylokokker, da de er meget ens morfologisk og fysiologisk (.ikipedia, Staph Aureus).
mit mål i dette eksperiment var at isolere, karakterisere og identificere en bakteriekoloni, der opstod fra en prøve taget fra min værelseskammerats næse. Jeg antog, at det ville være en bakterie, der ofte findes i næsehulen og sandsynligvis fra den førnævnte phyla, så det ville sandsynligvis gøre det bedst at leve i et aerobt, fugtigt og varmt miljø.
metoder:
Jeg valgte at prøve bakterier inde fra min værelseskammerats næse., For at prøve brugte jeg sterile bomuldspindler og stribede dem på TSA-plader. Jeg holdt pladen ved stuetemperatur i 7 dage og valgte derefter en koloni til at rense ved hjælp af metoden med ren kulturstrimmel. Jeg gentog denne proces tre gange for yderligere at rense kolonien. Når kulturen blev betragtet som ren nok, inokulerede jeg et skråt rør.
jeg udførte mange tests for at finde ud af kolonimorfologien og fysiologien., For at bestemme fysiologiske egenskaber ved kulturen, såsom celleform, arrangement, og om det var gram-positivt eller negativt, hvilket hjælper med at bestemme mikrobens cellevæg, udførte jeg en gram-plet. Jeg brugte en O .idase-teststrimmel og vand til at bestemme, om cytokrom C-O .idase var til stede, og udførte en katalase-test for at bestemme, om katalase var til stede. Jeg lavede også en væskethioglycolatest for at bestemme bakteriens iltklasse., Jeg voksede mine bakterier på en Eosin methylenblåt (EMB) plade for at se, om det kunne fermentere lactose, og om det kunne vokse med methylenblåt, som vælger for gramnegative bakterier. Jeg voksede det også i en MacConkey (MAC) plade for at se, om det kunne fermentere lactose, og om det kunne vokse med både krystalviolet og galdesalte for yderligere at bekræfte, om det var gram-negativt eller positivt. Jeg brugte en API-Strep-test til at bestemme flere af de sukkerarter, bakterierne kunne fermentere.
Jeg voksede min isoleret i Tryptisk soja bouillon (TSB) i en uge for at forberede mig på DNA-ekstraktion., Jeg ekstraherede DNA ‘ et ved hjælp af Po .ersoil DNA kit (fremstillet af .iagen) efter producentens anvisninger. Prøven blev derefter sekventeret ved hjælp af Illumina mise. – teknologien i UAFS DNA-Kernelaboratorium. Jeg brugte PATRIC soft .are til at udføre en metagenome binning og tildele en taksonomi til bakterierne.
resultater:
kolonien tog 16 dage at blive renset. Gram-pletten af denne mikrobe viste, at den er gram positiv, fordi den farvede lilla. Denne mikrobe er coccusformet og danner i tetrader. Kolonien dannes som en gul, skinnende rund klat., Katalase-og O .idasetestene kom negativt op, fordi katalasetesten ikke dannede bobler, og O .idasetesten så ikke en farveændring. O .idasetesten tester for at se, om mikroben indeholder cytokrom c-O .idase. Katalase-testen tester for at se, om mikroben indeholder katalase. Væsketestet thioglycallat viste, at bakterierne var en obligatorisk aerobe, fordi væksten var koncentreret øverst på røret i det lyserøde område. MacConkey-agaren viste meget lidt vækst og havde ikke en farveændring, hvilket indikerede, at mikroben var gram-positiv og ikke en fermenter., EMB-agaren viste ingen vækst eller farveændring, hvilket også indikerede, at mikroben var gram-positiv og en ikke-fermenter.
den taksonomiske tildeling af denne mikrobe var micrococcus luteus, fordi det var den eneste bin, PATRIC gav. Det havde 27,372 contigs i samling. Det har flere gener med antibiotikaresistens, herunder dihydropteroatsyntase, glycerophosphoryldiesterphosphodiesterase og SSU ribosomale proteiner.
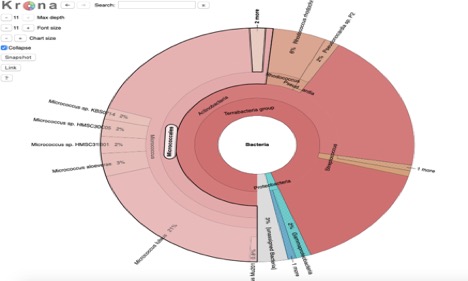 Figur 1. Krona diagram af mikrobe viser bakterielle klasser menes at være til stede i prøven.
Figur 1. Krona diagram af mikrobe viser bakterielle klasser menes at være til stede i prøven.
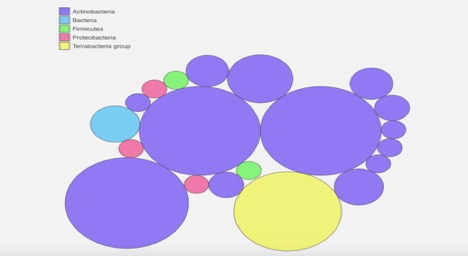 Figur 2., Kaiju metebserver metagenome binning analyse diagram. Det viser, at prøven indeholder bakterier fra Terrabacteria-gruppen. Det er for det meste Actinobacteria, men nogle Proteobacteria og Firmicules er også i prøven.
Figur 2., Kaiju metebserver metagenome binning analyse diagram. Det viser, at prøven indeholder bakterier fra Terrabacteria-gruppen. Det er for det meste Actinobacteria, men nogle Proteobacteria og Firmicules er også i prøven.
Kaiju metagenome binning viser, at mikrobe prøven ikke er helt ren (figur 2). Det viser, at det for det meste er Actinobacteria ,med nogle firmicules, og proteobacteria blandet i (figur 2). Dette matcher op med PATRIC metagenome binning som også viste nogle urenheder (Figur 1).,da mikroben er grampositiv, betyder det, at den har et stort peptidoglycan-lag og mangler et lipopolysaccharidlag. MacConkey agar er selektiv for gram-negativ, hvorfor min mikrobe ikke viste meget vækst på den, og fordi den ikke ændrede farver betyder det, at den ikke fermenterede laktosen. EMB-pladen er også selektiv for gram-negative bakterier, hvilket sandsynligvis er grunden til, at bakterierne ikke voksede på den. Mikrobens iltklasse, obligate aerobe, matcher de forudsigelser, jeg havde lavet om det, fordi bakterierne oprindeligt blev hentet i et næsebor., Microcikipedia siger også, at Micrococcus luteus er en obligatorisk aerobe, der sikkerhedskopierer, hvad mine resultater viser (2019).
o .idasetestresultaterne antyder, at mikroben ikke indeholder O .idase, på trods af hvad metagenom-binningstesten viste. Katalasetesten indikerede også, at mikroben ikke har katalase, på trods af at den metagenomiske binningstest antyder den. Disse uoverensstemmelser kan skyldes menneskelig fejl, uren kultur eller en gammel agarplade., API-teststrimlerne manglende resultater antyder, at jeg brugte den forkerte teststrimmel, jeg havde sandsynligvis brug for at bruge Staph-testen i stedet for Strep-testen, fordi Strep-testen er til, når katalase er fraværende, men der kunne have været katalase til stede. De modstridende resultater af metagenome-binning og katalase-testen påvirkede denne fejl. Jeg tror baseret på alle disse oplysninger, at min mikrobe er faktisk micrococcus luteus som foreslået af PATRIC metagenome binning test, og kronen (Figur 1).afslutningsvis var nogle af mine resultater inkonklusive og modstridende., Dette er sandsynligvis enten en årsag til menneskelig fejl, urene kulturer eller ikke at bruge agarplader, der er friske nok til testen. Jeg tror, at denne kultur var for det meste Micrococcus luteus baseret på Kaiju og metagenome arkivering resultater. Iltklassen og gram-positiviteten af mikroben stemmer også overens med Micrococcus luteus. I fremtiden arbejder med denne mikrobe, vil jeg nok gerne rense kulturen mere og gentage testene.
Bassis CM, AL Tang, VB Young, and MA Pynnonen (2014). Den nasale hulrum mikrobiota af raske voksne. Mikrobiom 2 (27).,
ra ,is m, and AK Ellis (2019). Næsens mikrobiom. Annaler for Allergi, Astma og Immunologi 122 (1): 17-24.
(2010) Hvad er mikrober? Institut for kvalitet og effektivitet i sundhedsvæsenet.